Python Pandas: astuces et fonctionnalités que vous ne connaissez peut-être pas
Pandas est une bibliothèque fondamentale pour l'analyse, le traitement des données et la science des données. C'est un énorme projet avec des tonnes d'options et de profondeur.
Ce didacticiel couvrira certaines capacités de Pandas moins utilisées mais idiomatiques qui confèrent à votre code une meilleure lisibilité, polyvalence et vitesse,à la la liste Buzzfeed.
Si vous vous sentez à l'aise avec les concepts de base de la bibliothèque Pandas de Python, nous espérons que vous trouverez une ou deux astuces dans cet article que vous n'avez pas rencontrées auparavant. (Si vous débutez avec la bibliothèque,10 Minutes to Pandas est un bon point de départ.)
Note: les exemples de cet article sont testés avec Pandas version 0.23.2 et Python 3.6.6. Cependant, ils devraient également être valides dans les anciennes versions.
1. Configurer les options et les paramètres au démarrage de l'interpréteur
Vous avez peut-être déjà utilisé le système richeoptions and settingsde Pandas.
C'est un énorme économiseur de productivité pour définir des options Pandas personnalisées au démarrage de l'interpréteur, surtout si vous travaillez dans un environnement de script. Vous pouvez utiliserpd.set_option() pour configurer à votre guise avec un fichier de démarragePython ouIPython.
Les options utilisent une notation par points telle quepd.set_option('display.max_colwidth', 25), qui se prête bien à un dictionnaire d'options imbriqué:
import pandas as pd
def start():
options = {
'display': {
'max_columns': None,
'max_colwidth': 25,
'expand_frame_repr': False, # Don't wrap to multiple pages
'max_rows': 14,
'max_seq_items': 50, # Max length of printed sequence
'precision': 4,
'show_dimensions': False
},
'mode': {
'chained_assignment': None # Controls SettingWithCopyWarning
}
}
for category, option in options.items():
for op, value in option.items():
pd.set_option(f'{category}.{op}', value) # Python 3.6+
if __name__ == '__main__':
start()
del start # Clean up namespace in the interpreterSi vous lancez une session d'interpréteur, vous verrez que tout dans le script de démarrage a été exécuté et que Pandas est importé automatiquement pour vous avec votre suite d'options:
>>>
>>> pd.__name__
'pandas'
>>> pd.get_option('display.max_rows')
14Utilisons des données sur lesabalone hébergés par le référentiel UCI Machine Learning pour illustrer le formatage qui a été défini dans le fichier de démarrage. Les données seront tronquées à 14 lignes avec 4 chiffres de précision pour les flottants:
>>>
>>> url = ('https://archive.ics.uci.edu/ml/'
... 'machine-learning-databases/abalone/abalone.data')
>>> cols = ['sex', 'length', 'diam', 'height', 'weight', 'rings']
>>> abalone = pd.read_csv(url, usecols=[0, 1, 2, 3, 4, 8], names=cols)
>>> abalone
sex length diam height weight rings
0 M 0.455 0.365 0.095 0.5140 15
1 M 0.350 0.265 0.090 0.2255 7
2 F 0.530 0.420 0.135 0.6770 9
3 M 0.440 0.365 0.125 0.5160 10
4 I 0.330 0.255 0.080 0.2050 7
5 I 0.425 0.300 0.095 0.3515 8
6 F 0.530 0.415 0.150 0.7775 20
# ...
4170 M 0.550 0.430 0.130 0.8395 10
4171 M 0.560 0.430 0.155 0.8675 8
4172 F 0.565 0.450 0.165 0.8870 11
4173 M 0.590 0.440 0.135 0.9660 10
4174 M 0.600 0.475 0.205 1.1760 9
4175 F 0.625 0.485 0.150 1.0945 10
4176 M 0.710 0.555 0.195 1.9485 12Vous verrez également cet ensemble de données s'afficher dans d'autres exemples ultérieurement.
2. Créez des structures de données de jouets avec le module de test Pandas
Le moduletesting de Pandas cache un certain nombre de fonctions pratiques pour créer rapidement des séries et des DataFrames quasi réalistes:
>>>
>>> import pandas.util.testing as tm
>>> tm.N, tm.K = 15, 3 # Module-level default rows/columns
>>> import numpy as np
>>> np.random.seed(444)
>>> tm.makeTimeDataFrame(freq='M').head()
A B C
2000-01-31 0.3574 -0.8804 0.2669
2000-02-29 0.3775 0.1526 -0.4803
2000-03-31 1.3823 0.2503 0.3008
2000-04-30 1.1755 0.0785 -0.1791
2000-05-31 -0.9393 -0.9039 1.1837
>>> tm.makeDataFrame().head()
A B C
nTLGGTiRHF -0.6228 0.6459 0.1251
WPBRn9jtsR -0.3187 -0.8091 1.1501
7B3wWfvuDA -1.9872 -1.0795 0.2987
yJ0BTjehH1 0.8802 0.7403 -1.2154
0luaYUYvy1 -0.9320 1.2912 -0.2907Il y en a environ 30, et vous pouvez voir la liste complète en appelantdir() sur l'objet module. Voici quelques-uns:
>>>
>>> [i for i in dir(tm) if i.startswith('make')]
['makeBoolIndex',
'makeCategoricalIndex',
'makeCustomDataframe',
'makeCustomIndex',
# ...,
'makeTimeSeries',
'makeTimedeltaIndex',
'makeUIntIndex',
'makeUnicodeIndex']Ceux-ci peuvent être utiles pour l'analyse comparative, le test d'assertions et l'expérimentation de méthodes Pandas que vous connaissez moins.
3. Profitez des méthodes d'accesseur
Peut-être avez-vous entendu parler du termeaccessor, qui ressemble un peu à un getter (bien que les getters et les setters soient rarement utilisés en Python). Pour nos besoins ici, vous pouvez considérer un accesseur Pandas comme une propriété qui sert d'interface à des méthodes supplémentaires.
Les séries Pandas en ont trois:
>>>
>>> pd.Series._accessors
{'cat', 'str', 'dt'}Oui, cette définition ci-dessus est une bouchée, alors examinons quelques exemples avant de discuter des internes.
.cat est pour les données catégorielles,.str est pour les données de chaîne (objet) et.dt est pour les données de type datetime. Commençons par.str: imaginez que vous ayez des données brutes de ville / état / ZIP dans un seul champ dans une série Pandas.
Les méthodes de chaîne Pandas sontvectorized, ce qui signifie qu'elles fonctionnent sur l'ensemble du tableau sans boucle for explicite:
>>>
>>> addr = pd.Series([
... 'Washington, D.C. 20003',
... 'Brooklyn, NY 11211-1755',
... 'Omaha, NE 68154',
... 'Pittsburgh, PA 15211'
... ])
>>> addr.str.upper()
0 WASHINGTON, D.C. 20003
1 BROOKLYN, NY 11211-1755
2 OMAHA, NE 68154
3 PITTSBURGH, PA 15211
dtype: object
>>> addr.str.count(r'\d') # 5 or 9-digit zip?
0 5
1 9
2 5
3 5
dtype: int64Pour un exemple plus complexe, supposons que vous souhaitiez séparer soigneusement les trois composants ville / état / ZIP en champs DataFrame.
Vous pouvez passer unregular expression à.str.extract() pour «extraire» des parties de chaque cellule de la série. Dans.str.extract(),.str est l'accesseur, et.str.extract() est une méthode d'accesseur:
>>>
>>> regex = (r'(?P[A-Za-z ]+), ' # One or more letters
... r'(?P[A-Z]{2}) ' # 2 capital letters
... r'(?P\d{5}(?:-\d{4})?)') # Optional 4-digit extension
...
>>> addr.str.replace('.', '').str.extract(regex)
city state zip
0 Washington DC 20003
1 Brooklyn NY 11211-1755
2 Omaha NE 68154
3 Pittsburgh PA 15211 Cela illustre également ce que l'on appelle le chaînage de méthodes, où.str.extract(regex) est appelé sur le résultat deaddr.str.replace('.', ''), ce qui nettoie l'utilisation des points pour obtenir une belle abréviation d'état à 2 caractères.
Il est utile d’en savoir un peu plus sur le fonctionnement de ces méthodes d’accesseurs en tant que raison motivante pour laquelle vous devriez les utiliser en premier lieu, plutôt que quelque chose commeaddr.apply(re.findall, ...).
Chaque accesseur est lui-même une classe Python authentique:
-
.strcorrespond àStringMethods. -
.dtcorrespond àCombinedDatetimelikeProperties. -
.catachemine versCategoricalAccessor.
Ces classes autonomes sont ensuite «attachées» à la classe Series à l'aide d'unCachedAccessor. C'est lorsque les classes sont enveloppées dansCachedAccessor qu'un peu de magie se produit.
CachedAccessor est inspiré d'une conception de «propriété mise en cache»: une propriété n'est calculée qu'une fois par instance, puis remplacée par un attribut ordinaire. Il le fait en surchargeant lahttps://docs.python.org/reference/datamodel.html#object.get [méthode.__get__()], qui fait partie du protocole de descripteur de Python.
Note: si vous souhaitez en savoir plus sur le fonctionnement interne de cela, consultez lesPython Descriptor HOWTO etthis post sur la conception de la propriété mise en cache. Python 3 a également introduitfunctools.lru_cache(), qui offre des fonctionnalités similaires. Il y a des exemples partout de ce modèle, comme dans le packageaiohttp.
Le deuxième accesseur,.dt, est pour les données de type datetime. Il appartient techniquement auxDatetimeIndex de Pandas, et s'il est appelé sur une série, il est d'abord converti enDatetimeIndex:
>>>
>>> daterng = pd.Series(pd.date_range('2017', periods=9, freq='Q'))
>>> daterng
0 2017-03-31
1 2017-06-30
2 2017-09-30
3 2017-12-31
4 2018-03-31
5 2018-06-30
6 2018-09-30
7 2018-12-31
8 2019-03-31
dtype: datetime64[ns]
>>> daterng.dt.day_name()
0 Friday
1 Friday
2 Saturday
3 Sunday
4 Saturday
5 Saturday
6 Sunday
7 Monday
8 Sunday
dtype: object
>>> # Second-half of year only
>>> daterng[daterng.dt.quarter > 2]
2 2017-09-30
3 2017-12-31
6 2018-09-30
7 2018-12-31
dtype: datetime64[ns]
>>> daterng[daterng.dt.is_year_end]
3 2017-12-31
7 2018-12-31
dtype: datetime64[ns]Le troisième accesseur,.cat, concerne uniquement les données catégorielles, que vous verrez bientôt dans sesown section.
4. Créer un DatetimeIndex à partir des colonnes de composants
En parlant de données de type datetime, comme dansdaterng ci-dessus, il est possible de créer un PandasDatetimeIndex à partir de plusieurs colonnes de composants qui forment ensemble une date ou une date / heure:
>>>
>>> from itertools import product
>>> datecols = ['year', 'month', 'day']
>>> df = pd.DataFrame(list(product([2017, 2016], [1, 2], [1, 2, 3])),
... columns=datecols)
>>> df['data'] = np.random.randn(len(df))
>>> df
year month day data
0 2017 1 1 -0.0767
1 2017 1 2 -1.2798
2 2017 1 3 0.4032
3 2017 2 1 1.2377
4 2017 2 2 -0.2060
5 2017 2 3 0.6187
6 2016 1 1 2.3786
7 2016 1 2 -0.4730
8 2016 1 3 -2.1505
9 2016 2 1 -0.6340
10 2016 2 2 0.7964
11 2016 2 3 0.0005
>>> df.index = pd.to_datetime(df[datecols])
>>> df.head()
year month day data
2017-01-01 2017 1 1 -0.0767
2017-01-02 2017 1 2 -1.2798
2017-01-03 2017 1 3 0.4032
2017-02-01 2017 2 1 1.2377
2017-02-02 2017 2 2 -0.2060Enfin, vous pouvez supprimer les anciennes colonnes individuelles et les convertir en série:
>>>
>>> df = df.drop(datecols, axis=1).squeeze()
>>> df.head()
2017-01-01 -0.0767
2017-01-02 -1.2798
2017-01-03 0.4032
2017-02-01 1.2377
2017-02-02 -0.2060
Name: data, dtype: float64
>>> df.index.dtype_str
'datetime64[ns]L'intuition derrière la transmission d'un DataFrame est qu'un DataFrame ressemble à un dictionnaire Python où les noms de colonne sont des clés et les colonnes individuelles (Series) sont les valeurs du dictionnaire. C’est pourquoipd.to_datetime(df[datecols].to_dict(orient='list')) fonctionnerait également dans ce cas. Cela reflète la construction desdatetime.datetime de Python, où vous passez des arguments de mot-clé tels quedatetime.datetime(year=2000, month=1, day=15, hour=10).
5. Utilisez des données catégoriques pour économiser du temps et de l'espace
Une caractéristique puissante de Pandas est son dtypeCategorical.
Même si vous ne travaillez pas toujours avec des gigaoctets de données dans la RAM, vous avez probablement rencontré des cas où les opérations simples sur un grand DataFrame semblent se bloquer pendant plus de quelques secondes.
Le dtype de Pandasobjectest souvent un excellent candidat pour la conversion en données de catégorie. (object est un conteneur pour Pythonstr, les types de données hétérogènes ou les «autres» types.) Les chaînes occupent une quantité importante d'espace en mémoire:
>>>
>>> colors = pd.Series([
... 'periwinkle',
... 'mint green',
... 'burnt orange',
... 'periwinkle',
... 'burnt orange',
... 'rose',
... 'rose',
... 'mint green',
... 'rose',
... 'navy'
... ])
...
>>> import sys
>>> colors.apply(sys.getsizeof)
0 59
1 59
2 61
3 59
4 61
5 53
6 53
7 59
8 53
9 53
dtype: int64Note: J'ai utilisésys.getsizeof() pour afficher la mémoire occupée par chaque valeur individuelle de la série. Gardez à l'esprit que ce sont des objets Python qui ont des frais généraux en premier lieu. (sys.getsizeof('') renverra 49 octets.)
Il existe égalementcolors.memory_usage(), qui résume l'utilisation de la mémoire et repose sur l'attribut.nbytes du tableau NumPy sous-jacent. Ne vous embourbez pas trop dans ces détails: ce qui est important, c'est l'utilisation relative de la mémoire qui résulte de la conversion de type, comme vous le verrez ensuite.
Maintenant, que se passerait-il si nous pouvions prendre les couleurs uniques ci-dessus et les associer à un entier moins encombrant? Voici une implémentation naïve de cela:
>>>
>>> mapper = {v: k for k, v in enumerate(colors.unique())}
>>> mapper
{'periwinkle': 0, 'mint green': 1, 'burnt orange': 2, 'rose': 3, 'navy': 4}
>>> as_int = colors.map(mapper)
>>> as_int
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int64
>>> as_int.apply(sys.getsizeof)
0 24
1 28
2 28
3 24
4 28
5 28
6 28
7 28
8 28
9 28
dtype: int64Note: Une autre façon de faire la même chose est avec lespd.factorize(colors) de Pandas:
>>>
>>> pd.factorize(colors)[0]
array([0, 1, 2, 0, 2, 3, 3, 1, 3, 4])Dans les deux cas, vous codez l'objet en tant que type énuméré (variable catégorielle).
Vous remarquerez immédiatement que l'utilisation de la mémoire est à peu près divisée par deux par rapport à l'utilisation des chaînes complètes avec le dtypeobject.
Plus tôt dans la section suraccessors, j'ai mentionné l'accesseur.cat (catégorique). Ce qui précède avecmapper est une illustration approximative de ce qui se passe en interne avec le dtypeCategorical de Pandas:
«L'utilisation de la mémoire d'un
Categoricalest proportionnelle au nombre de catégories plus la longueur des données. En revanche, un dtypeobjectest une constante multipliée par la longueur des données. » (Source)
Encolors ci-dessus, vous avez un rapport de 2 valeurs pour chaque valeur unique (catégorie):
>>>
>>> len(colors) / colors.nunique()
2.0En conséquence, les économies de mémoire résultant de la conversion enCategorical sont bonnes, mais pas excellentes:
>>>
>>> # Not a huge space-saver to encode as Categorical
>>> colors.memory_usage(index=False, deep=True)
650
>>> colors.astype('category').memory_usage(index=False, deep=True)
495Cependant, si vous expliquez la proportion ci-dessus, avec beaucoup de données et peu de valeurs uniques (pensez aux données sur les données démographiques ou les résultats des tests alphabétiques), la réduction de la mémoire requise est plus de 10 fois:
>>>
>>> manycolors = colors.repeat(10)
>>> len(manycolors) / manycolors.nunique() # Much greater than 2.0x
20.0
>>> manycolors.memory_usage(index=False, deep=True)
6500
>>> manycolors.astype('category').memory_usage(index=False, deep=True)
585Un bonus est que l'efficacité de calcul est également augmentée: pour lesSeries catégoriques, les opérations de chaîneare performed on the .cat.categories attribute plutôt que sur chaque élément d'origine desSeries.
En d'autres termes, l'opération est effectuée une fois par catégorie unique et les résultats sont mappés sur les valeurs. Les données catégorielles ont un accesseur.cat qui est une fenêtre sur les attributs et les méthodes de manipulation des catégories:
>>>
>>> ccolors = colors.astype('category')
>>> ccolors.cat.categories
Index(['burnt orange', 'mint green', 'navy', 'periwinkle', 'rose'], dtype='object')En fait, vous pouvez reproduire quelque chose de similaire à l'exemple ci-dessus que vous avez fait manuellement:
>>>
>>> ccolors.cat.codes
0 3
1 1
2 0
3 3
4 0
5 4
6 4
7 1
8 4
9 2
dtype: int8Tout ce que vous devez faire pour imiter exactement la sortie manuelle précédente est de réorganiser les codes:
>>>
>>> ccolors.cat.reorder_categories(mapper).cat.codes
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int8Notez que le dtype estint8 de NumPy, un8-bit signed integer qui peut prendre des valeurs de -127 à 128. (Un seul octet est nécessaire pour représenter une valeur en mémoire. Lesints signés 64 bits seraient exagérés en termes d'utilisation de la mémoire.) Notre exemple grossier a abouti à des donnéesint64 par défaut, alors que Pandas est suffisamment intelligent pour réduire les données catégorielles au plus petit type numérique possible. .
La plupart des attributs de.cat sont liés à l'affichage et à la manipulation des catégories sous-jacentes elles-mêmes:
>>>
>>> [i for i in dir(ccolors.cat) if not i.startswith('_')]
['add_categories',
'as_ordered',
'as_unordered',
'categories',
'codes',
'ordered',
'remove_categories',
'remove_unused_categories',
'rename_categories',
'reorder_categories',
'set_categories']Il y a cependant quelques mises en garde. Les données catégorielles sont généralement moins flexibles. Par exemple, si vous insérez des valeurs précédemment invisibles, vous devez d'abord ajouter cette valeur à un conteneur.categories:
>>>
>>> ccolors.iloc[5] = 'a new color'
# ...
ValueError: Cannot setitem on a Categorical with a new category,
set the categories first
>>> ccolors = ccolors.cat.add_categories(['a new color'])
>>> ccolors.iloc[5] = 'a new color' # No more ValueErrorSi vous prévoyez de définir des valeurs ou de remodeler des données plutôt que de dériver de nouveaux calculs, les typesCategorical peuvent être moins agiles.
6. Introspecter Groupby Objects via Iteration
Lorsque vous appelezdf.groupby('x'), les objets Pandasgroupby résultants peuvent être un peu opaques. Cet objet est instancié paresseusement et n'a pas de représentation significative en lui-même.
Vous pouvez démontrer avec l'ensemble de données abalone deexample 1:
>>>
>>> abalone['ring_quartile'] = pd.qcut(abalone.rings, q=4, labels=range(1, 5))
>>> grouped = abalone.groupby('ring_quartile')
>>> grouped
D'accord, vous avez maintenant un objetgroupby, mais qu'est-ce que c'est, et comment le voir?
Avant d'appeler quelque chose commegrouped.apply(func), vous pouvez profiter du fait que les objetsgroupby sont itérables:
>>>
>>> help(grouped.__iter__)
Groupby iterator
Returns
-------
Generator yielding sequence of (name, subsetted object)
for each groupChaque «élément» produit pargrouped.__iter__() est un tuple de(name, subsetted object), oùname est la valeur de la colonne sur laquelle vous groupez etsubsetted object est un DataFrame qui est un sous-ensemble du DataFrame d'origine basé sur la condition de regroupement que vous spécifiez. Autrement dit, les données sont fragmentées par groupe:
>>>
>>> for idx, frame in grouped:
... print(f'Ring quartile: {idx}')
... print('-' * 16)
... print(frame.nlargest(3, 'weight'), end='\n\n')
...
Ring quartile: 1
----------------
sex length diam height weight rings ring_quartile
2619 M 0.690 0.540 0.185 1.7100 8 1
1044 M 0.690 0.525 0.175 1.7005 8 1
1026 M 0.645 0.520 0.175 1.5610 8 1
Ring quartile: 2
----------------
sex length diam height weight rings ring_quartile
2811 M 0.725 0.57 0.190 2.3305 9 2
1426 F 0.745 0.57 0.215 2.2500 9 2
1821 F 0.720 0.55 0.195 2.0730 9 2
Ring quartile: 3
----------------
sex length diam height weight rings ring_quartile
1209 F 0.780 0.63 0.215 2.657 11 3
1051 F 0.735 0.60 0.220 2.555 11 3
3715 M 0.780 0.60 0.210 2.548 11 3
Ring quartile: 4
----------------
sex length diam height weight rings ring_quartile
891 M 0.730 0.595 0.23 2.8255 17 4
1763 M 0.775 0.630 0.25 2.7795 12 4
165 M 0.725 0.570 0.19 2.5500 14 4De même, un objetgroupby a également.groups et un groupe-getter,.get_group():
>>>
>>> grouped.groups.keys()
dict_keys([1, 2, 3, 4])
>>> grouped.get_group(2).head()
sex length diam height weight rings ring_quartile
2 F 0.530 0.420 0.135 0.6770 9 2
8 M 0.475 0.370 0.125 0.5095 9 2
19 M 0.450 0.320 0.100 0.3810 9 2
23 F 0.550 0.415 0.135 0.7635 9 2
39 M 0.355 0.290 0.090 0.3275 9 2Cela peut vous aider à être un peu plus sûr que l'opération que vous effectuez est celle que vous souhaitez:
>>>
>>> grouped['height', 'weight'].agg(['mean', 'median'])
height weight
mean median mean median
ring_quartile
1 0.1066 0.105 0.4324 0.3685
2 0.1427 0.145 0.8520 0.8440
3 0.1572 0.155 1.0669 1.0645
4 0.1648 0.165 1.1149 1.0655Quel que soit le calcul que vous effectuez surgrouped, qu'il s'agisse d'une seule méthode Pandas ou d'une fonction personnalisée, chacune de ces «sous-trames» est passée une par une comme argument de cet appelable. C'est de là que vient le terme «fractionner-appliquer-combiner»: décomposer les données par groupes, effectuer un calcul par groupe et recombiner de manière agrégée.
Si vous ne parvenez pas à visualiser exactement à quoi ressembleront réellement les groupes, il suffit de les parcourir et d'en imprimer quelques-uns pour être extrêmement utile.
7. Utilisez cette astuce de mappage pour le regroupement des membres
Supposons que vous ayez une série et une «table de mappage» correspondante où chaque valeur appartient à un groupe multi-membres, ou à aucun groupe du tout:
>>>
>>> countries = pd.Series([
... 'United States',
... 'Canada',
... 'Mexico',
... 'Belgium',
... 'United Kingdom',
... 'Thailand'
... ])
...
>>> groups = {
... 'North America': ('United States', 'Canada', 'Mexico', 'Greenland'),
... 'Europe': ('France', 'Germany', 'United Kingdom', 'Belgium')
... }En d'autres termes, vous devez mappercountries au résultat suivant:
>>>
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: objectCe dont vous avez besoin ici, c’est une fonction similaire auxpd.cut() de Pandas, mais pour le regroupement basé sur l’appartenance catégorielle. Vous pouvez utiliserpd.Series.map(), que vous avez déjà vu dansexample #5, pour imiter ceci:
from typing import Any
def membership_map(s: pd.Series, groups: dict,
fillvalue: Any=-1) -> pd.Series:
# Reverse & expand the dictionary key-value pairs
groups = {x: k for k, v in groups.items() for x in v}
return s.map(groups).fillna(fillvalue)Cela devrait être beaucoup plus rapide qu'une boucle Python imbriquée viagroups pour chaque pays encountries.
Voici un essai routier:
>>>
>>> membership_map(countries, groups, fillvalue='other')
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: objectDécrivons ce qui se passe ici. (Sidenote: c'est un excellent endroit pour entrer dans la portée d'une fonction avec le débogueur de Python,pdb, pour inspecter quelles variables sont locales à la fonction.)
L'objectif est de mapper chaque groupe engroups sur un entier. Cependant,Series.map() ne reconnaîtra pas'ab' - il a besoin de la version éclatée avec chaque caractère de chaque groupe mappé à un entier. Voici ce que font lesdictionary comprehension:
>>>
>>> groups = dict(enumerate(('ab', 'cd', 'xyz')))
>>> {x: k for k, v in groups.items() for x in v}
{'a': 0, 'b': 0, 'c': 1, 'd': 1, 'x': 2, 'y': 2, 'z': 2}Ce dictionnaire peut être passé às.map() pour mapper ou «traduire» ses valeurs en leurs index de groupe correspondants.
8. Comprendre comment Pandas utilise les opérateurs booléens
Vous connaissez peut-être lesoperator precedence de Python, oùand,not etor ont une priorité inférieure à celle des opérateurs arithmétiques tels que<,<= ,>,>=,!= et==. Considérez les deux instructions ci-dessous, où< et> ont une priorité plus élevée que l'opérateurand:
>>>
>>> # Evaluates to "False and True"
>>> 4 < 3 and 5 > 4
False
>>> # Evaluates to 4 < 5 > 4
>>> 4 < (3 and 5) > 4
TrueNote: il n’est pas spécifiquement lié aux pandas, mais3 and 5 s’évalue à5 en raison de l’évaluation des courts-circuits:
"La valeur de retour d'un opérateur de court-circuit est le dernier argument évalué." (Source)
Pandas (et NumPy, sur lesquels Pandas est construit) n'utilise pasand,or ounot. Au lieu de cela, il utilise respectivement&,| et~, qui sont des opérateurs binaires Python normaux et authentiques.
Ces opérateurs ne sont pas «inventés» par les Pandas. Au contraire,&,| et~ sont des opérateurs Python intégrés valides qui ont une priorité plus élevée (plutôt que inférieure) que les opérateurs arithmétiques. (Pandas remplace les méthodes dunder comme.__ror__() qui correspondent à l'opérateur|.) Pour sacrifier certains détails, vous pouvez considérer «au niveau du bit» comme «par élément» en ce qui concerne Pandas et NumPy:
>>>
>>> pd.Series([True, True, False]) & pd.Series([True, False, False])
0 True
1 False
2 False
dtype: boolIl est utile de comprendre ce concept dans son intégralité. Disons que vous avez une série semblable à une gamme:
>>>
>>> s = pd.Series(range(10))Je suppose que vous avez peut-être vu cette exception soulevée à un moment donné:
>>>
>>> s % 2 == 0 & s > 3
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().Qu'est-ce qu'il se passe ici? Il est utile de lier progressivement l'expression entre parenthèses, expliquant comment Python développe cette expression étape par étape:
s % 2 == 0 & s > 3 # Same as above, original expression
(s % 2) == 0 & s > 3 # Modulo is most tightly binding here
(s % 2) == (0 & s) > 3 # Bitwise-and is second-most-binding
(s % 2) == (0 & s) and (0 & s) > 3 # Expand the statement
((s % 2) == (0 & s)) and ((0 & s) > 3) # The `and` operator is least-bindingL'expressions % 2 == 0 & s > 3 équivaut à (ou est traitée comme)((s % 2) == (0 & s)) and ((0 & s) > 3). C'est ce qu'on appelleexpansion:x < y <= z équivaut àx < y and y <= z.
Bon, arrêtons-nous maintenant, et revenons à Pandas. Vous avez deux séries Pandas que nous appelleronsleft etright:
>>>
>>> left = (s % 2) == (0 & s)
>>> right = (0 & s) > 3
>>> left and right # This will raise the same ValueErrorVous savez qu'une instruction de la formeleft and right est une valeur de vérité testant à la foisleft etright, comme dans ce qui suit:
>>>
>>> bool(left) and bool(right)Le problème est que les développeurs Pandas n’établissent pas intentionnellement une valeur de vérité (véracité) pour une série entière. Une série est-elle vraie ou fausse? Qui sait? Le résultat est ambigu:
>>>
>>> bool(s)
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().La seule comparaison qui a du sens est une comparaison élémentaire. C’est pourquoi, si un opérateur arithmétique est impliqué,you’ll need parentheses:
>>>
>>> (s % 2 == 0) & (s > 3)
0 False
1 False
2 False
3 False
4 True
5 False
6 True
7 False
8 True
9 False
dtype: boolEn bref, si vous voyez lesValueError ci-dessus apparaître avec une indexation booléenne, la première chose que vous devriez probablement chercher à faire est de mettre les parenthèses nécessaires.
9. Charger des données depuis le presse-papiers
Il est courant de devoir transférer des données d’un endroit comme Excel ouSublime Text vers une structure de données Pandas. Idéalement, vous voulez le faire sans passer par l'étape intermédiaire de l'enregistrement des données dans un fichier et ensuite de la lecture du fichier dans Pandas.
Vous pouvez charger des DataFrames à partir du tampon de données du presse-papiers de votre ordinateur avecpd.read_clipboard(). Ses arguments de mot-clé sont transmis àpd.read_table().
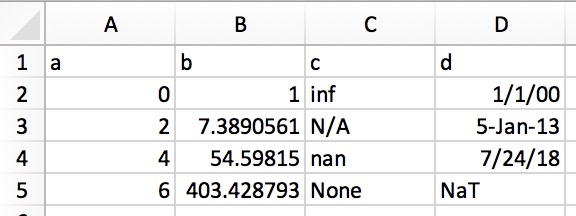
Cela vous permet de copier du texte structuré directement vers un DataFrame ou une série. Dans Excel, les données ressembleraient à ceci:
Sa représentation en texte brut (par exemple, dans un éditeur de texte) ressemblerait à ceci:
a b c d 0 1 inf 1/1/00 2 7.389056099 N/A 5-Jan-13 4 54.59815003 nan 7/24/18 6 403.4287935 None NaT
Mettez simplement en surbrillance et copiez le texte brut ci-dessus et appelezpd.read_clipboard():
>>>
>>> df = pd.read_clipboard(na_values=[None], parse_dates=['d'])
>>> df
a b c d
0 0 1.0000 inf 2000-01-01
1 2 7.3891 NaN 2013-01-05
2 4 54.5982 NaN 2018-07-24
3 6 403.4288 NaN NaT
>>> df.dtypes
a int64
b float64
c float64
d datetime64[ns]
dtype: object10. Écrire des objets Pandas directement au format compressé
Celui-ci est court et doux pour compléter la liste. À partir de la version 0.21.0 de Pandas, vous pouvez écrire des objets Pandas directement en compression gzip, bz2, zip ou xz, plutôt que de stocker le fichier non compressé en mémoire et de le convertir. Voici un exemple utilisant les donnéesabalone detrick #1:
abalone.to_json('df.json.gz', orient='records',
lines=True, compression='gzip')Dans ce cas, la différence de taille est de 11,6x:
>>>
>>> import os.path
>>> abalone.to_json('df.json', orient='records', lines=True)
>>> os.path.getsize('df.json') / os.path.getsize('df.json.gz')
11.603035760226396Vous souhaitez ajouter à cette liste? Faites le nous savoir
J'espère que vous avez pu prendre quelques astuces utiles dans cette liste pour améliorer la lisibilité, la polyvalence et les performances de votre code Pandas.
Si vous avez quelque chose dans votre manche qui n’est pas couvert ici, veuillez laisser une suggestion dans les commentaires ou sous forme deGitHub Gist. Nous ajouterons volontiers à cette liste et donnerons le crédit qui lui revient.