Python Pandas: Tricks und Funktionen, die Sie möglicherweise nicht kennen
Pandas ist eine grundlegende Bibliothek für Analytik, Datenverarbeitung und Datenwissenschaft. Es ist ein riesiges Projekt mit einer Menge Optionalität und Tiefe.
Dieses Tutorial behandelt einige weniger genutzte, aber idiomatische Pandas-Funktionen, die Ihrem Code eine bessere Lesbarkeit, Vielseitigkeit und Geschwindigkeit verleihen, à la the Buzzfeed listicle.
Wenn Sie mit den Kernkonzepten der Pandas-Bibliothek von Python vertraut sind, finden Sie in diesem Artikel hoffentlich ein oder zwei Tricks, auf die Sie zuvor noch nicht gestoßen sind. (Wenn Sie gerade erst mit der Bibliothek beginnen, ist 10 Minuten bis Pandas ein guter Anfang.)
*Hinweis* : Die Beispiele in diesem Artikel wurden mit Pandas Version 0.23.2 und Python 3.6.6 getestet. Sie sollten jedoch auch in älteren Versionen gültig sein.
1. Konfigurieren Sie Optionen und Einstellungen beim Interpreter-Start
Möglicherweise sind Sie zuvor auf das umfangreiche options and settings -System von Pandas gestoßen.
Es ist eine enorme Produktivitätsersparnis, beim Start des Interpreters benutzerdefinierte Pandas-Optionen festzulegen, insbesondere wenn Sie in einer Skriptumgebung arbeiten. Sie können + pd.set_option () + verwenden, um nach Herzenslust mit Python oder https:/zu konfigurieren Startdatei/ipython.readthedocs.io/en/stable/interactive/tutorial.html#startup-files[IPython].
Die Optionen verwenden eine Punktnotation wie "+ pd.set_option (" display.max_colwidth ", 25) +", die sich gut für ein verschachteltes Wörterbuch von Optionen eignet:
import pandas as pd
def start():
options = {
'display': {
'max_columns': None,
'max_colwidth': 25,
'expand_frame_repr': False, # Don't wrap to multiple pages
'max_rows': 14,
'max_seq_items': 50, # Max length of printed sequence
'precision': 4,
'show_dimensions': False
},
'mode': {
'chained_assignment': None # Controls SettingWithCopyWarning
}
}
for category, option in options.items():
for op, value in option.items():
pd.set_option(f'{category}.{op}', value) # Python 3.6+
if __name__ == '__main__':
start()
del start # Clean up namespace in the interpreterWenn Sie eine Dolmetschersitzung starten, sehen Sie, dass alles im Startskript ausgeführt wurde und Pandas automatisch mit Ihrer Optionssuite für Sie importiert wird:
>>>
>>> pd.__name__
'pandas'
>>> pd.get_option('display.max_rows')
14Verwenden wir einige Daten auf abalone, die vom UCI Machine Learning Repository gehostet werden, um die in der Startdatei festgelegte Formatierung zu demonstrieren. Die Daten werden bei 14 Zeilen mit 4 Stellen Genauigkeit für Floats abgeschnitten:
>>>
>>> url = ('https://archive.ics.uci.edu/ml/'
... 'machine-learning-databases/abalone/abalone.data')
>>> cols = ['sex', 'length', 'diam', 'height', 'weight', 'rings']
>>> abalone = pd.read_csv(url, usecols=[0, 1, 2, 3, 4, 8], names=cols)
>>> abalone
sex length diam height weight rings
0 M 0.455 0.365 0.095 0.5140 15
1 M 0.350 0.265 0.090 0.2255 7
2 F 0.530 0.420 0.135 0.6770 9
3 M 0.440 0.365 0.125 0.5160 10
4 I 0.330 0.255 0.080 0.2050 7
5 I 0.425 0.300 0.095 0.3515 8
6 F 0.530 0.415 0.150 0.7775 20
# ...
4170 M 0.550 0.430 0.130 0.8395 10
4171 M 0.560 0.430 0.155 0.8675 8
4172 F 0.565 0.450 0.165 0.8870 11
4173 M 0.590 0.440 0.135 0.9660 10
4174 M 0.600 0.475 0.205 1.1760 9
4175 F 0.625 0.485 0.150 1.0945 10
4176 M 0.710 0.555 0.195 1.9485 12Dieser Datensatz wird später auch in anderen Beispielen angezeigt.
2. Erstellen Sie Spielzeugdatenstrukturen mit dem Testmodul von Pandas
Versteckt im Pandas-Modul https://github.com/pandas-dev/pandas/blob/master/pandas/util/testing.py [+ testing +] befinden sich eine Reihe praktischer Funktionen, mit denen Sie schnell quasi-realistisch erstellen können Serien- und DataFrames:
>>>
>>> import pandas.util.testing as tm
>>> tm.N, tm.K = 15, 3 # Module-level default rows/columns
>>> import numpy as np
>>> np.random.seed(444)
>>> tm.makeTimeDataFrame(freq='M').head()
A B C
2000-01-31 0.3574 -0.8804 0.2669
2000-02-29 0.3775 0.1526 -0.4803
2000-03-31 1.3823 0.2503 0.3008
2000-04-30 1.1755 0.0785 -0.1791
2000-05-31 -0.9393 -0.9039 1.1837
>>> tm.makeDataFrame().head()
A B C
nTLGGTiRHF -0.6228 0.6459 0.1251
WPBRn9jtsR -0.3187 -0.8091 1.1501
7B3wWfvuDA -1.9872 -1.0795 0.2987
yJ0BTjehH1 0.8802 0.7403 -1.2154
0luaYUYvy1 -0.9320 1.2912 -0.2907Es gibt ungefähr 30 davon, und Sie können die vollständige Liste anzeigen, indem Sie "+ dir () +" für das Modulobjekt aufrufen. Hier sind ein paar:
>>>
>>> [i for i in dir(tm) if i.startswith('make')]
['makeBoolIndex',
'makeCategoricalIndex',
'makeCustomDataframe',
'makeCustomIndex',
# ...,
'makeTimeSeries',
'makeTimedeltaIndex',
'makeUIntIndex',
'makeUnicodeIndex']Diese können nützlich sein, um Benchmarking durchzuführen, Behauptungen zu testen und mit Pandas-Methoden zu experimentieren, mit denen Sie weniger vertraut sind.
3. Nutzen Sie die Accessor-Methoden
Vielleicht haben Sie schon von dem Begriff Accessor gehört, der einem Getter ähnelt (obwohl Getter und Setter in Python selten verwendet werden). Für unsere Zwecke hier können Sie sich einen Pandas-Accessor als eine Eigenschaft vorstellen, die als Schnittstelle zu zusätzlichen Methoden dient.
Die Pandas-Serie hat drei davon:
>>>
>>> pd.Series._accessors
{'cat', 'str', 'dt'}Ja, diese Definition oben ist ein Mund voll. Schauen wir uns also einige Beispiele an, bevor wir die Interna diskutieren.
+ .cat + steht für kategoriale Daten, + .str + steht für String- (Objekt-) Daten und + .dt + steht für datetime-ähnliche Daten. Beginnen wir mit + .str +: Stellen Sie sich vor, Sie haben einige Rohdaten zu Stadt/Bundesland/Postleitzahl als einzelnes Feld innerhalb einer Pandas-Serie.
Pandas-String-Methoden sind vectorized, was bedeutet, dass sie auf dem gesamten Array ohne explizite for-Schleife ausgeführt werden:
>>>
>>> addr = pd.Series([
... 'Washington, D.C. 20003',
... 'Brooklyn, NY 11211-1755',
... 'Omaha, NE 68154',
... 'Pittsburgh, PA 15211'
... ])
>>> addr.str.upper()
0 WASHINGTON, D.C. 20003
1 BROOKLYN, NY 11211-1755
2 OMAHA, NE 68154
3 PITTSBURGH, PA 15211
dtype: object
>>> addr.str.count(r'\d') # 5 or 9-digit zip?
0 5
1 9
2 5
3 5
dtype: int64Angenommen, Sie möchten die drei Komponenten Stadt/Bundesland/Postleitzahl sauber in DataFrame-Felder aufteilen.
Sie können einen regulären Ausdruck an + .str.extract () + übergeben, um Teile jeder Zelle in der Serie zu "extrahieren". In + .str.extract () + ist + .str + der Accessor und + .str.extract () + ist eine Accessormethode:
>>>
>>> regex = (r'(?P<city>[A-Za-z ]+), ' # One or more letters
... r'(?P<state>[A-Z]{2}) ' # 2 capital letters
... r'(?P<zip>\d{5}(?:-\d{4})?)') # Optional 4-digit extension
...
>>> addr.str.replace('.', '').str.extract(regex)
city state zip
0 Washington DC 20003
1 Brooklyn NY 11211-1755
2 Omaha NE 68154
3 Pittsburgh PA 15211Dies zeigt auch die sogenannte Methodenverkettung, bei der "+ .str.extract (regex) " für das Ergebnis von " addr.str.replace (". ",") + "Aufgerufen wird, das bereinigt Verwendung von Punkten, um eine schöne 2-stellige Statusabkürzung zu erhalten.
Es ist hilfreich, ein wenig darüber zu wissen, wie diese Accessor-Methoden als Motivationsgrund dafür dienen, warum Sie sie überhaupt verwenden sollten, anstatt so etwas wie "+ addr.apply (re.findall, …) +".
Jeder Accessor ist selbst eine echte Python-Klasse:
-
+ .str +wird https://github.com/pandas-dev/pandas/blob/3e4839301fc2927646889b194c9eb41c62b76bda/pandas/core/strings.py#L1766 [+ StringMethods +] zugeordnet. -
+ .dt +wird https://github.com/pandas-dev/pandas/blob/3e4839301fc2927646889b194c9eb41c62b76bda/pandas/core/indexes/accessors.py#L306 [`+ CombinedDatetimelikeProperties +] zugeordnet. -
+ .cat +Routen zu https://github.com/pandas-dev/pandas/blob/3e4839301fc2927646889b194c9eb41c62b76bda/pandas/core/arrays/categorical.py#L2356 [+ CategoricalAccessor +].
Diese eigenständigen Klassen werden dann mithilfe von https://github.com/pandas-dev/pandas/blob/master/pandas/core/accessor.py [+ CachedAccessor +] an die Series-Klasse „angehängt“. Wenn die Klassen in + CachedAccessor + eingeschlossen sind, passiert ein bisschen Magie.
+ CachedAccessor + ist von einem "zwischengespeicherten Eigenschaft" -Design inspiriert: Eine Eigenschaft wird nur einmal pro Instanz berechnet und dann durch ein gewöhnliches Attribut ersetzt. Dazu wird die https://docs.python.org/reference/datamodel.html#object.get [+ . get () + Methode] überladen, die Teil des Python-Deskriptorprotokolls ist.
*Hinweis* : Wenn Sie mehr über die Interna erfahren möchten, wie dies funktioniert, lesen Sie https://docs.python.org/howto/descriptor.html[Python Descriptor HOWTO] und https://www.pydanny .com/cached-property.html [dieser Beitrag] zum Design der zwischengespeicherten Eigenschaft. Python 3 führte auch https://docs.python.org/library/functools.html#functools.lru_cache [`+ functools.lru_cache () +`] ein, das ähnliche Funktionen bietet. Es gibt überall Beispiele für dieses Muster, z. B. im Paket https://github.com/aio-libs/aiohttp/blob/master/aiohttp/_helpers.pyx [`+ aiohttp +`].
Der zweite Accessor, "+ .dt ", ist für datetime-ähnliche Daten. Es gehört technisch zu Pandas '' + DatetimeIndex + '' und wenn es für eine Serie aufgerufen wird, wird es zuerst in einen ' DatetimeIndex +' konvertiert:
>>>
>>> daterng = pd.Series(pd.date_range('2017', periods=9, freq='Q'))
>>> daterng
0 2017-03-31
1 2017-06-30
2 2017-09-30
3 2017-12-31
4 2018-03-31
5 2018-06-30
6 2018-09-30
7 2018-12-31
8 2019-03-31
dtype: datetime64[ns]
>>> daterng.dt.day_name()
0 Friday
1 Friday
2 Saturday
3 Sunday
4 Saturday
5 Saturday
6 Sunday
7 Monday
8 Sunday
dtype: object
>>> # Second-half of year only
>>> daterng[daterng.dt.quarter > 2]
2 2017-09-30
3 2017-12-31
6 2018-09-30
7 2018-12-31
dtype: datetime64[ns]
>>> daterng[daterng.dt.is_year_end]
3 2017-12-31
7 2018-12-31
dtype: datetime64[ns]Der dritte Accessor, "+ .cat +", ist nur für kategoriale Daten vorgesehen, die Sie in Kürze in seinem Link sehen werden: # 5 - Verwenden Sie kategoriale Daten, um Zeit und Platz zu sparen [eigener Abschnitt] .
4. Erstellen Sie einen DatetimeIndex aus Komponentenspalten
Wenn Sie von datetime-ähnlichen Daten sprechen, wie in + daterng + oben, ist es möglich, einen Pandas + DatetimeIndex + aus mehreren Komponentenspalten zu erstellen, die zusammen ein Datum oder eine datetime bilden:
>>>
>>> from itertools import product
>>> datecols = ['year', 'month', 'day']
>>> df = pd.DataFrame(list(product([2017, 2016], [1, 2], [1, 2, 3])),
... columns=datecols)
>>> df['data'] = np.random.randn(len(df))
>>> df
year month day data
0 2017 1 1 -0.0767
1 2017 1 2 -1.2798
2 2017 1 3 0.4032
3 2017 2 1 1.2377
4 2017 2 2 -0.2060
5 2017 2 3 0.6187
6 2016 1 1 2.3786
7 2016 1 2 -0.4730
8 2016 1 3 -2.1505
9 2016 2 1 -0.6340
10 2016 2 2 0.7964
11 2016 2 3 0.0005
>>> df.index = pd.to_datetime(df[datecols])
>>> df.head()
year month day data
2017-01-01 2017 1 1 -0.0767
2017-01-02 2017 1 2 -1.2798
2017-01-03 2017 1 3 0.4032
2017-02-01 2017 2 1 1.2377
2017-02-02 2017 2 2 -0.2060Schließlich können Sie die alten einzelnen Spalten löschen und in eine Serie konvertieren:
>>>
>>> df = df.drop(datecols, axis=1).squeeze()
>>> df.head()
2017-01-01 -0.0767
2017-01-02 -1.2798
2017-01-03 0.4032
2017-02-01 1.2377
2017-02-02 -0.2060
Name: data, dtype: float64
>>> df.index.dtype_str
'datetime64[ns]Die Intuition hinter der Übergabe eines DataFrame besteht darin, dass ein DataFrame einem Python-Wörterbuch ähnelt, bei dem die Spaltennamen Schlüssel und die einzelnen Spalten (Serien) die Wörterbuchwerte sind. Aus diesem Grund würde "+ pd.to_datetime (df [datecols] .to_dict (orient =" list ")) " auch in diesem Fall funktionieren. Dies spiegelt die Konstruktion von Pythons " datetime.datetime " wider, bei der Sie Schlüsselwortargumente wie " datetime.datetime (Jahr = 2000, Monat = 1, Tag = 15, Stunde = 10) +" übergeben.
5. Verwenden Sie kategoriale Daten, um Zeit und Raum zu sparen
Eine mächtige Pandas-Funktion ist der Typ "+ Categorical +".
Selbst wenn Sie nicht immer mit Gigabyte Daten im RAM arbeiten, werden Sie wahrscheinlich auf Fälle stoßen, in denen unkomplizierte Vorgänge auf einem großen DataFrame länger als ein paar Sekunden hängen bleiben.
Pandas + object + dtype ist oft ein großartiger Kandidat für die Konvertierung in Kategoriedaten. (+ object + ist ein Container für Python + str +, heterogene Datentypen oder "andere" Typen.) Strings nehmen viel Speicherplatz ein:
>>>
>>> colors = pd.Series([
... 'periwinkle',
... 'mint green',
... 'burnt orange',
... 'periwinkle',
... 'burnt orange',
... 'rose',
... 'rose',
... 'mint green',
... 'rose',
... 'navy'
... ])
...
>>> import sys
>>> colors.apply(sys.getsizeof)
0 59
1 59
2 61
3 59
4 61
5 53
6 53
7 59
8 53
9 53
dtype: int64*Hinweis:* Ich habe `+ sys.getsizeof () +` verwendet, um den Speicher anzuzeigen, der von jedem einzelnen Wert in der Serie belegt wird. Beachten Sie, dass dies Python-Objekte sind, die in erster Linie einen gewissen Overhead haben. (`+ sys.getsizeof ('') +` gibt 49 Bytes zurück.)
Es gibt auch + color.memory_usage () +, das die Speichernutzung zusammenfasst und sich auf das Attribut + .nbytes + des zugrunde liegenden NumPy-Arrays stützt. Lassen Sie sich in diesen Details nicht zu sehr einklemmen: Wichtig ist die relative Speichernutzung, die sich aus der Typkonvertierung ergibt, wie Sie als Nächstes sehen werden.
Was wäre, wenn wir die oben genannten einzigartigen Farben auf eine weniger platzraubende Ganzzahl abbilden könnten? Hier ist eine naive Implementierung davon:
>>>
>>> mapper = {v: k for k, v in enumerate(colors.unique())}
>>> mapper
{'periwinkle': 0, 'mint green': 1, 'burnt orange': 2, 'rose': 3, 'navy': 4}
>>> as_int = colors.map(mapper)
>>> as_int
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int64
>>> as_int.apply(sys.getsizeof)
0 24
1 28
2 28
3 24
4 28
5 28
6 28
7 28
8 28
9 28
dtype: int64*Hinweis* : Eine andere Möglichkeit, dasselbe zu tun, ist Pandas '+ pd.factorize (Farben) +':
>>>
>>> pd.factorize(colors)[0]
array([0, 1, 2, 0, 2, 3, 3, 1, 3, 4])In beiden Fällen codieren Sie das Objekt als Aufzählungstyp (kategoriale Variable).
Sie werden sofort feststellen, dass sich die Speichernutzung im Vergleich zur Verwendung der vollständigen Zeichenfolgen mit "+ object +" dtype nur halbiert.
Zu Beginn des Abschnitts über den Link: # 3-nutzen-von-Accessor-Methoden [Accessoren] habe ich den (kategorialen) Accessor "+ .cat " erwähnt. Das obige mit " Mapper " ist eine grobe Illustration dessen, was intern mit Pandas " Categorical +" dtype passiert:
_
„Die Speichernutzung eines` + Categorical + ist proportional zur Anzahl der Kategorien plus der Länge der Daten. Im Gegensatz dazu ist ein `+ Objekt + d-Typ eine Konstante mal die Länge der Daten. ” (Source)
_
In + Farben + oben haben Sie ein Verhältnis von 2 Werten für jeden eindeutigen Wert (Kategorie):
>>>
>>> len(colors)/colors.nunique()
2.0Infolgedessen ist die Speicherersparnis durch die Konvertierung in "+ Categorical +" gut, aber nicht großartig:
>>>
>>> # Not a huge space-saver to encode as Categorical
>>> colors.memory_usage(index=False, deep=True)
650
>>> colors.astype('category').memory_usage(index=False, deep=True)
495Wenn Sie jedoch den oben genannten Anteil mit vielen Daten und wenigen eindeutigen Werten ausblasen (denken Sie an Daten zu demografischen Merkmalen oder alphabetischen Testergebnissen), beträgt der Speicherbedarf mehr als das Zehnfache:
>>>
>>> manycolors = colors.repeat(10)
>>> len(manycolors)/manycolors.nunique() # Much greater than 2.0x
20.0
>>> manycolors.memory_usage(index=False, deep=True)
6500
>>> manycolors.astype('category').memory_usage(index=False, deep=True)
585Ein Bonus ist, dass die Recheneffizienz ebenfalls gesteigert wird: Für kategoriale "+ Series " werden die Zeichenfolgenoperationen https://pandas.pydata.org/pandas-docs/stable/text.html[ auf der ` .cat ausgeführt. Kategorien + Attribut] anstatt auf jedem Originalelement der + Serie + `.
Mit anderen Worten, die Operation wird einmal pro eindeutiger Kategorie ausgeführt, und die Ergebnisse werden wieder den Werten zugeordnet. Kategoriale Daten haben einen "+ .cat +" - Accessor, der ein Fenster mit Attributen und Methoden zum Bearbeiten der Kategorien darstellt:
>>>
>>> ccolors = colors.astype('category')
>>> ccolors.cat.categories
Index(['burnt orange', 'mint green', 'navy', 'periwinkle', 'rose'], dtype='object')Tatsächlich können Sie etwas Ähnliches wie im obigen Beispiel reproduzieren, das Sie manuell erstellt haben:
>>>
>>> ccolors.cat.codes
0 3
1 1
2 0
3 3
4 0
5 4
6 4
7 1
8 4
9 2
dtype: int8Alles, was Sie tun müssen, um die frühere manuelle Ausgabe genau nachzuahmen, ist die Neuordnung der Codes:
>>>
>>> ccolors.cat.reorder_categories(mapper).cat.codes
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int8Beachten Sie, dass der dtype NumPys + int8 + ist, eine 8-bit signierte Ganzzahl, die Werte von annehmen kann -127 bis 128. (Es wird nur ein einziges Byte benötigt, um einen Wert im Speicher darzustellen. 64-Bit-signierte + Ints + wären in Bezug auf die Speichernutzung übertrieben.) Unser grob behauenes Beispiel führte standardmäßig zu + int64 + Daten, während Pandas klug genug ist, kategoriale Daten auf den kleinstmöglichen numerischen Typ herunterzuschieben.
Die meisten Attribute für "+ .cat +" beziehen sich auf das Anzeigen und Bearbeiten der zugrunde liegenden Kategorien selbst:
>>>
>>> [i for i in dir(ccolors.cat) if not i.startswith('_')]
['add_categories',
'as_ordered',
'as_unordered',
'categories',
'codes',
'ordered',
'remove_categories',
'remove_unused_categories',
'rename_categories',
'reorder_categories',
'set_categories']Es gibt jedoch einige Einschränkungen. Kategoriale Daten sind im Allgemeinen weniger flexibel. Wenn Sie beispielsweise zuvor nicht sichtbare Werte einfügen, müssen Sie diesen Wert zuerst einem Container "+ .categories +" hinzufügen:
>>>
>>> ccolors.iloc[5] = 'a new color'
# ...
ValueError: Cannot setitem on a Categorical with a new category,
set the categories first
>>> ccolors = ccolors.cat.add_categories(['a new color'])
>>> ccolors.iloc[5] = 'a new color' # No more ValueErrorWenn Sie vorhaben, Werte festzulegen oder Daten neu zu formen, anstatt neue Berechnungen abzuleiten, sind + kategoriale + Typen möglicherweise weniger flink.
6. Introspect Groupby Objects via Iteration
Wenn Sie "+ df.groupby (" x ") " aufrufen, können die resultierenden Pandas " groupby +" Objekte etwas undurchsichtig sein. Dieses Objekt wird träge instanziiert und hat selbst keine aussagekräftige Darstellung.
Sie können dies mit dem Abalone-Datensatz unter folgendem Link demonstrieren: # 1-configure-options-settings-at-interpreter-startup [Beispiel 1]:
>>>
>>> abalone['ring_quartile'] = pd.qcut(abalone.rings, q=4, labels=range(1, 5))
>>> grouped = abalone.groupby('ring_quartile')
>>> grouped
<pandas.core.groupby.groupby.DataFrameGroupBy object at 0x11c1169b0>Okay, jetzt hast du ein + groupby + Objekt, aber was ist das für ein Ding und wie sehe ich es?
Bevor Sie so etwas wie "+ grouped.apply (func) " aufrufen, können Sie die Tatsache nutzen, dass " groupby +" - Objekte iterierbar sind:
>>>
>>> help(grouped.__iter__)
Groupby iterator
Returns
-------
Generator yielding sequence of (name, subsetted object)
for each groupJedes "Ding", das von "+ gruppiert . iter () " ergibt, ist ein Tupel von " (Name, untergeordnetes Objekt) ", wobei " Name +" der Wert der Spalte ist, in der Sie gruppieren, und " + subsetted object + `ist ein DataFrame, der eine Teilmenge des ursprünglichen DataFrame ist, basierend auf der von Ihnen angegebenen Gruppierungsbedingung. Das heißt, die Daten werden nach Gruppen aufgeteilt:
>>>
>>> for idx, frame in grouped:
... print(f'Ring quartile: {idx}')
... print('-' * 16)
... print(frame.nlargest(3, 'weight'), end='\n\n')
...
Ring quartile: 1
----------------
sex length diam height weight rings ring_quartile
2619 M 0.690 0.540 0.185 1.7100 8 1
1044 M 0.690 0.525 0.175 1.7005 8 1
1026 M 0.645 0.520 0.175 1.5610 8 1
Ring quartile: 2
----------------
sex length diam height weight rings ring_quartile
2811 M 0.725 0.57 0.190 2.3305 9 2
1426 F 0.745 0.57 0.215 2.2500 9 2
1821 F 0.720 0.55 0.195 2.0730 9 2
Ring quartile: 3
----------------
sex length diam height weight rings ring_quartile
1209 F 0.780 0.63 0.215 2.657 11 3
1051 F 0.735 0.60 0.220 2.555 11 3
3715 M 0.780 0.60 0.210 2.548 11 3
Ring quartile: 4
----------------
sex length diam height weight rings ring_quartile
891 M 0.730 0.595 0.23 2.8255 17 4
1763 M 0.775 0.630 0.25 2.7795 12 4
165 M 0.725 0.570 0.19 2.5500 14 4In ähnlicher Weise hat ein + groupby + Objekt auch` + .groups + und einen Group-Getter, + .get_group () + `:
>>>
>>> grouped.groups.keys()
dict_keys([1, 2, 3, 4])
>>> grouped.get_group(2).head()
sex length diam height weight rings ring_quartile
2 F 0.530 0.420 0.135 0.6770 9 2
8 M 0.475 0.370 0.125 0.5095 9 2
19 M 0.450 0.320 0.100 0.3810 9 2
23 F 0.550 0.415 0.135 0.7635 9 2
39 M 0.355 0.290 0.090 0.3275 9 2Dies kann Ihnen helfen, ein wenig sicherer zu sein, dass die von Ihnen ausgeführte Operation die gewünschte ist:
>>>
>>> grouped['height', 'weight'].agg(['mean', 'median'])
height weight
mean median mean median
ring_quartile
1 0.1066 0.105 0.4324 0.3685
2 0.1427 0.145 0.8520 0.8440
3 0.1572 0.155 1.0669 1.0645
4 0.1648 0.165 1.1149 1.0655Unabhängig davon, welche Berechnung Sie für "+ gruppiert +" durchführen, sei es eine einzelne Pandas-Methode oder eine benutzerdefinierte Funktion, wird jeder dieser "Unterrahmen" einzeln als Argument an diesen Aufrufer übergeben. Hier kommt der Begriff „Split-Apply-Combine“ her: Teilen Sie die Daten nach Gruppen auf, führen Sie eine Berechnung pro Gruppe durch und kombinieren Sie sie auf aggregierte Weise neu.
Wenn Sie Probleme haben, genau zu visualisieren, wie die Gruppen tatsächlich aussehen, kann es äußerst nützlich sein, sie einfach zu durchlaufen und einige zu drucken.
7. Verwenden Sie diesen Mapping-Trick für das Binning von Mitgliedschaften
Angenommen, Sie haben eine Serie und eine entsprechende "Zuordnungstabelle", in der jeder Wert zu einer Gruppe mit mehreren Mitgliedern oder zu keiner Gruppe gehört:
>>>
>>> countries = pd.Series([
... 'United States',
... 'Canada',
... 'Mexico',
... 'Belgium',
... 'United Kingdom',
... 'Thailand'
... ])
...
>>> groups = {
... 'North America': ('United States', 'Canada', 'Mexico', 'Greenland'),
... 'Europe': ('France', 'Germany', 'United Kingdom', 'Belgium')
... }Mit anderen Worten, Sie müssen "+ Länder +" dem folgenden Ergebnis zuordnen:
>>>
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: objectWas Sie hier benötigen, ist eine Funktion ähnlich der von Pandas "+ pd.cut () ", jedoch zum Binning basierend auf einer kategorialen Mitgliedschaft. Sie können ` pd.Series.map () +` verwenden, das Sie bereits im folgenden Link gesehen haben: # 5-Verwenden Sie kategoriale Daten, um Zeit und Platz zu sparen [Beispiel # 5], um nachzuahmen Dies:
from typing import Any
def membership_map(s: pd.Series, groups: dict,
fillvalue: Any=-1) -> pd.Series:
# Reverse & expand the dictionary key-value pairs
groups = {x: k for k, v in groups.items() for x in v}
return s.map(groups).fillna(fillvalue)Dies sollte deutlich schneller sein als eine verschachtelte Python-Schleife durch "+ Gruppen " für jedes Land in " Ländern +".
Hier ist eine Probefahrt:
>>>
>>> membership_map(countries, groups, fillvalue='other')
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: objectLassen Sie uns zusammenfassen, was hier los ist. (Nebenbemerkung: Dies ist ein großartiger Ort, um mit dem Python-Debugger + pdb + in den Funktionsumfang einer Funktion einzusteigen und zu überprüfen, welche Variablen für die Funktion lokal sind.)
Das Ziel ist es, jede Gruppe in "+ Gruppen " einer ganzen Zahl zuzuordnen. ` Series.map () ` erkennt jedoch nicht ` 'ab' +` - es wird die ausgebrochene Version benötigt, wobei jedes Zeichen aus jeder Gruppe einer Ganzzahl zugeordnet ist. Dies ist, was das dictionary-Verständnis tut:
>>>
>>> groups = dict(enumerate(('ab', 'cd', 'xyz')))
>>> {x: k for k, v in groups.items() for x in v}
{'a': 0, 'b': 0, 'c': 1, 'd': 1, 'x': 2, 'y': 2, 'z': 2}Dieses Wörterbuch kann an + s.map () + übergeben werden, um seine Werte auf die entsprechenden Gruppenindizes abzubilden oder zu „übersetzen“.
8. Verstehen, wie Pandas Boolesche Operatoren verwendet
Möglicherweise kennen Sie Pythons operator priority, wobei + und +, + not + und + oder + eine niedrigere Priorität haben als arithmetische Operatoren wie "+ <", " ⇐ ", "> ", "> = ", "! = " und " == ". Betrachten Sie die beiden folgenden Anweisungen, wobei ` <` und `> ` eine höhere Priorität haben als die Operatoren ` und +`:
>>>
>>> # Evaluates to "False and True"
>>> 4 < 3 and 5 > 4
False
>>> # Evaluates to 4 < 5 > 4
>>> 4 < (3 and 5) > 4
True*Hinweis* : Es handelt sich nicht speziell um Pandas, aber "+3 und 5 +" ergibt aufgrund der Kurzschlussbewertung "+ 5 +":
_ "Der Rückgabewert eines Kurzschlussoperators ist das zuletzt bewertete Argument." (Source) _
Pandas (und NumPy, auf dem Pandas basiert) verwenden nicht "+ und ", " oder " oder " nicht ". Stattdessen werden " & ", " | " bzw. " ~ +" verwendet, die normale, gutgläubige bitweise Python-Operatoren sind.
Diese Operatoren werden von Pandas nicht „erfunden“. Vielmehr sind "+ & ", " | " und " ~ " gültige integrierte Python-Operatoren, die eine höhere (und keine niedrigere) Priorität haben als arithmetische Operatoren. (Pandas überschreibt Dunder-Methoden wie " . ror () ", die dem Operator " | +" zugeordnet sind.) Um einige Details zu opfern, können Sie sich "bitweise" als "elementweise" vorstellen, wenn es sich um Pandas und NumPy handelt:
>>>
>>> pd.Series([True, True, False]) & pd.Series([True, False, False])
0 True
1 False
2 False
dtype: boolEs lohnt sich, dieses Konzept vollständig zu verstehen. Nehmen wir an, Sie haben eine Range-ähnliche Serie:
>>>
>>> s = pd.Series(range(10))Ich würde vermuten, dass Sie diese Ausnahme irgendwann gesehen haben:
>>>
>>> s % 2 == 0 & s > 3
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().Was passiert hier? Es ist hilfreich, den Ausdruck schrittweise in Klammern zu binden und darzulegen, wie Python diesen Ausdruck Schritt für Schritt erweitert:
s % 2 == 0 & s > 3 # Same as above, original expression
(s % 2) == 0 & s > 3 # Modulo is most tightly binding here
(s % 2) == (0 & s) > 3 # Bitwise-and is second-most-binding
(s % 2) == (0 & s) and (0 & s) > 3 # Expand the statement
((s % 2) == (0 & s)) and ((0 & s) > 3) # The `and` operator is least-bindingDer Ausdruck "+ s% 2 == 0 & s> 3 " entspricht (oder wird behandelt als) " s% 2) == (0 & s und ((0 & s)> 3 ) + . Dies wird als https://docs.python.org/reference/expressions.html#comparisons[expansion bezeichnet: `+ x <y ⇐ z + entspricht + x <y und y ⇐ z +.
Okay, jetzt hör dort auf und lass uns das zurück zu Pandas-speak bringen. Sie haben zwei Pandas-Serien, die wir "+ left " und " right +" nennen:
>>>
>>> left = (s % 2) == (0 & s)
>>> right = (0 & s) > 3
>>> left and right # This will raise the same ValueErrorSie wissen, dass eine Aussage der Form "+ links und rechts " ein Wahrheitswert ist, der sowohl " links " als auch " rechts +" testet, wie im Folgenden:
>>>
>>> bool(left) and bool(right)Das Problem ist, dass Pandas-Entwickler absichtlich keinen Wahrheitswert (Wahrhaftigkeit) für eine ganze Serie festlegen. Ist eine Serie wahr oder falsch? Wer weiß? Das Ergebnis ist nicht eindeutig:
>>>
>>> bool(s)
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().Der einzige sinnvolle Vergleich ist ein elementweiser Vergleich. Wenn ein arithmetischer Operator beteiligt ist, benötigen Sie daher https://pandas.pydata.org/pandas-docs/stable/indexing.html#boolean-indexing [Sie benötigen Klammern]:
>>>
>>> (s % 2 == 0) & (s > 3)
0 False
1 False
2 False
3 False
4 True
5 False
6 True
7 False
8 True
9 False
dtype: boolKurz gesagt, wenn das obige + ValueError + mit boolescher Indizierung angezeigt wird, sollten Sie wahrscheinlich zuerst einige erforderliche Klammern einstreuen.
9. Laden Sie Daten aus der Zwischenablage
Es ist eine häufige Situation, Daten von einem Ort wie Excel oder Sublime Text auf einen Pandas übertragen zu müssen Datenstruktur. Idealerweise möchten Sie dies tun, ohne den Zwischenschritt des Speicherns der Daten in einer Datei und des anschließenden Einlesens der Datei in Pandas zu durchlaufen.
Sie können DataFrames aus dem Datenpuffer der Zwischenablage Ihres Computers mit https://pandas.pydata.org/pandas-docs/stable/generated/pandas.read_clipboard.html [+ pd.read_clipboard () +] laden. Die Schlüsselwortargumente werden an https://pandas.pydata.org/pandas-docs/stable/generated/pandas.read_table.html [+ pd.read_table () +] weitergeleitet.
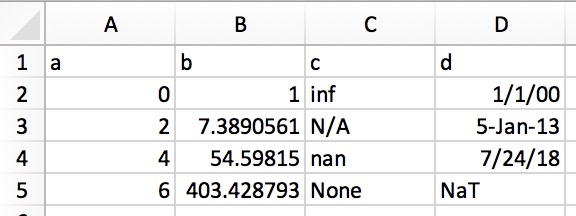
Auf diese Weise können Sie strukturierten Text direkt in einen DataFrame oder eine Serie kopieren. In Excel würden die Daten ungefähr so aussehen:
Die Klartextdarstellung (z. B. in einem Texteditor) würde folgendermaßen aussehen:
a b c d 0 1 inf 1/1/00 2 7.389056099 N/A 5-Jan-13 4 54.59815003 nan 7/24/18 6 403.4287935 None NaT
Markieren und kopieren Sie einfach den obigen Klartext und rufen Sie + pd.read_clipboard () + auf:
>>>
>>> df = pd.read_clipboard(na_values=[None], parse_dates=['d'])
>>> df
a b c d
0 0 1.0000 inf 2000-01-01
1 2 7.3891 NaN 2013-01-05
2 4 54.5982 NaN 2018-07-24
3 6 403.4288 NaN NaT
>>> df.dtypes
a int64
b float64
c float64
d datetime64[ns]
dtype: object10. Schreiben Sie Pandas-Objekte direkt in das komprimierte Format
Dieser ist kurz und bündig, um die Liste abzurunden. Ab Pandas Version 0.21.0 können Sie Pandas-Objekte direkt in die Komprimierung von gzip, bz2, zip oder xz schreiben, anstatt die unkomprimierte Datei im Speicher zu speichern und zu konvertieren. Hier ist ein Beispiel mit den Daten "+ abalone +" von link: # 1-configure-options-settings-at-interpreter-startup [Trick # 1]:
abalone.to_json('df.json.gz', orient='records',
lines=True, compression='gzip')In diesem Fall beträgt der Größenunterschied 11,6x:
>>>
>>> import os.path
>>> abalone.to_json('df.json', orient='records', lines=True)
>>> os.path.getsize('df.json')/os.path.getsize('df.json.gz')
11.603035760226396Möchten Sie dieser Liste hinzufügen? Lass uns wissen
Hoffentlich konnten Sie einige nützliche Tricks aus dieser Liste auswählen, um Ihrem Pandas-Code eine bessere Lesbarkeit, Vielseitigkeit und Leistung zu verleihen.
Wenn Sie etwas im Ärmel haben, das hier nicht behandelt wird, hinterlassen Sie bitte einen Vorschlag in den Kommentaren oder als GitHub Gist. Wir werden diese Liste gerne ergänzen und dort gutschreiben, wo sie fällig ist.